GRASS (por su siglas en inglés, the Geographic Resources Analysis and Support System, http://grass.itc.it) es un Sistema de Información Geografica (SIG) gratuito y de código abierto, integrado con un sistema de procesamiento de imágenes y subsistemas de visualización. GRASS permite trabajar con imágenes raster y vectorial. Tiene interfaces paraPostgreSQL, MySQL, DBF y bases de datos conectadas con ODBC. Además puede ser conectado a UMN/MapServer, R, gstat, Matlab, Octave, Povray y otros software.
Hasta hace poco siempre había trabajado con otros software comerciales como ArcGIS, Idrisi, PCI o ERDAS. GRASS siempre me había parecido muy complicado y poco amigable. Sin embargo, sus características como software libre y gratuito lo convierten en una herramienta muy versátil y potente, por lo que merece la pena hacer el esfuerzo de aprendizaje. En esta entrada explico como empezar a trabajar en GRASS y los conceptos más básicos para aquellos que, como yo hasta ayer, quieran dar el salto pero no acaban de animarse.
Instalación y manuales
GRASS está disponible desde el ITC-irst en Italia (http://grass.itc.it). GRASS está diseñado para correr en varios ambientes UNIX, tales como GNU/Linux, SUN-Solaris, Irix o MacOS X. También puede correr bajo MSWindows/NT/2000/XP con Cygwin (un simulador de Linux), aunque posiblemente las próximas versiones de GRASS se puedan correr bajo Windows directamente. Libros, tutoriales, manuales, cursos en línea y mas documentación se encuentra listada en el 'Proyecto para la Documentación de GRASS' (http://grass.itc.it/gdp). En español, hay al menos un par de tutoriales de introducción a GRASS (http://mpa.itc.it/markus/osg05/). Ambos son traducciones de un tutorial de Markus Neteler para un taller de trabajo y no difieren apenas en cuanto a contenidos entre sí. En mi opininión, estos tutoriales no son particularmente buenos, pero por lo menos sirven para entender algunos conceptos básicos del funcionamiento de GRASS, por lo que merece la pena echarles un vistazo (muchos de los contenidos de esta entrada han sido extraidos de estos tutoriales).
Estructura de los proyectos en GRASS
Cuando alguien está familiarizado con el uso de SIG y sensores remotos pero nunca antes había trabajado con GRASS, lo más importante es saber cómo se estructuran los proyectos y cómo abrir una sesión en GRASS. Esto es lo que normalmente más cuesta entender. La información de GRASS se encuentra almacenada en un directorio conocido como 'database' (también llamado 'GISDBASE') . Este directorio se debe de crear antes de empezar a trabajar con GRASS (yo lo he llamado 'home/luis/grassdata'). Dentro de este 'database', los proyectos están organizados por áreas de proyectos en subdirectorios llamados 'locations'. Un 'location' está definido por un sistema de coordenadas, una proyección cartográfica y unas fronteras geográficas. Los subdirectorios y archivos que definen un 'location' son creados automáticamente cuando GRASS es iniciado la primera vez con un nuevo 'location' (esto es lo que vamos a hacer un poco más abajo). Cada 'location' puede tener múltiples 'mapsets'. Un motivo para mantener múltiples 'mapsets' es almacenar mapas relacionados a algún aspecto específico del proyecto o subregiones. Otro motivo es permitir el trabajo simultáneo de usuarios a capas almacenadas dentro del el mismo 'location', por ejemplo, equipos trabajando en elmismo proyecto. Para trabajos en equipo, una base de datos de GRASS centralizada puede ser creada en un sistema de archivos de red (por ejemplo, NFS). Además de acceder el propio 'mapset', cada usuario puede leer capas de otros 'mapsets', aunque cada usuario solo puede modificar o borrar las capas en su propio 'mapset'.
Cuando se crea un nuevo 'location', GRASS automáticamente crea un 'mapset' especial llamado PERMANENT donde la información principal del proyecto puede ser almacenada. Datos en el 'mapset' PERMANENT pueden ser solamente agregados, modificados o borrados por el propietario del 'mapset' PERMANENT. Sin embargo, los datos pueden ser consultados, analizados y copiados por otros usuarios a sus respectivos 'mapsets'. El 'mapset' PERMANENT es útil para entregar información general (ejemplo, un modelo de elevación digital), accesible pero protegida contra escritura de todos los usuarios que se encuentran trabajando en el mismo 'location' del propietario de la base de datos. En principio si no se tiene pensado trabajar en equipos con varios usuarios y con una gran cantidad de información, se puede trabajar directamente en el 'mapset' PERMANENT.
Primeros pasos en GRASS: Cómo crear un nuevo 'location'
Una gran diferencia entre GRASS y otros SIG es que GRASS requiere los parámetros de la proyección antes de que el usuario pueda trabajar en un 'location'. La ventaja de esto es que las cosas quedan bien definidas y se evita un desorden con la combinación de proyecciones, cosa que en mi experiencia es bastante común cuando se trabaja con otros software que no siguen esta estructura de proyectos. Para crear un nuevo 'location' primero tenemos que abrir GRASS. En Linux (yo trabajo actualmente con Ubuntu Intrepid Ibex 8.10) hay que ir a la terminal (konsole) y escribir:
grass63
o simplemente:
grass
Esto abrira la ventana de bienvenida de GRASS.
 Lo primero que hay que hacer es seleccionar el 'database' donde se va a guardar el nuevo 'location'. Este directorio se ha tenido que crear antes de empezar a trabajar en GRASS. Ahora selecciona el botón 'Projection values' para crear un nuevo 'location' (Define new location with...), el cual te llevará a una nueva ventana de texto.
Lo primero que hay que hacer es seleccionar el 'database' donde se va a guardar el nuevo 'location'. Este directorio se ha tenido que crear antes de empezar a trabajar en GRASS. Ahora selecciona el botón 'Projection values' para crear un nuevo 'location' (Define new location with...), el cual te llevará a una nueva ventana de texto.
 En la pantalla ingresa un nuevo nombre para el 'location' (no puede contener espacios en blanco) y luego continua presionando la tecla ESC y la tecla RETURN de tu teclado. Te aparecerá otra ventana de texto diciendo que esa 'location' no existe, cuáles son las 'locations' disponibles y preguntándote si quieres crear una nueva 'location' con el nombre asignado. A esto último se dice que sí y se pulsa RETURN.
En la pantalla ingresa un nuevo nombre para el 'location' (no puede contener espacios en blanco) y luego continua presionando la tecla ESC y la tecla RETURN de tu teclado. Te aparecerá otra ventana de texto diciendo que esa 'location' no existe, cuáles son las 'locations' disponibles y preguntándote si quieres crear una nueva 'location' con el nombre asignado. A esto último se dice que sí y se pulsa RETURN.
 Esto te lleva a otra ventana de texto en dónde te explican la información que vas a necesitar para crear un nuevo 'location' (sistema de coordenadas, zona UTM y parámetros de la proyección, coordenadas del área de trabajo, etc). Te preguntará si tienes toda esa información. Se dice que sí y se pulsa RETURN.
Esto te lleva a otra ventana de texto en dónde te explican la información que vas a necesitar para crear un nuevo 'location' (sistema de coordenadas, zona UTM y parámetros de la proyección, coordenadas del área de trabajo, etc). Te preguntará si tienes toda esa información. Se dice que sí y se pulsa RETURN.
 Una nueva ventana te pedirá que definas el sistema de coordenadas para tu 'location'. En mi caso defino C (UTM) y pulso RETURN.
Una nueva ventana te pedirá que definas el sistema de coordenadas para tu 'location'. En mi caso defino C (UTM) y pulso RETURN.
 Lo siguiente es ingresar una pequeña descripción o título para el 'location'. En esta misma ventana te pregunta si quieres especificar un datum. Por defecto se responde que sí. Se pulsa RETURN para continuar.
Lo siguiente es ingresar una pequeña descripción o título para el 'location'. En esta misma ventana te pregunta si quieres especificar un datum. Por defecto se responde que sí. Se pulsa RETURN para continuar.
 Para especificar el datum se puede pulsar 'list' y te viene una lista de todos los datum disponibles. Para salir de este listado se pulsa 'q'. Se escribe el nombre del datum y se pulsa RETURN.
Para especificar el datum se puede pulsar 'list' y te viene una lista de todos los datum disponibles. Para salir de este listado se pulsa 'q'. Se escribe el nombre del datum y se pulsa RETURN.
 El último paso es definir las coordenadas del área de estudio. En mi caso por ejemplo, las hice coincidir con las del modelo digital de elevaciones con el que voy a trabajar. En otros casos pueden ser más o menos amplias y abarcar toda una región geográfica como Sierra Nevada, Andalucía o la Península Ibérica. Hay que tener en cuenta que esta es la región que define el 'location', pero que luego es posible redefinir el área de estudio en distintos 'mapset' dentro de un 'location'. Igualmente se pueden redefinir los parámetros de un 'location' una vez que ha sido creado.
El último paso es definir las coordenadas del área de estudio. En mi caso por ejemplo, las hice coincidir con las del modelo digital de elevaciones con el que voy a trabajar. En otros casos pueden ser más o menos amplias y abarcar toda una región geográfica como Sierra Nevada, Andalucía o la Península Ibérica. Hay que tener en cuenta que esta es la región que define el 'location', pero que luego es posible redefinir el área de estudio en distintos 'mapset' dentro de un 'location'. Igualmente se pueden redefinir los parámetros de un 'location' una vez que ha sido creado.
 Finalmente se pide que se defina la resolución de los píxeles. Una cosa buena de GRASS es que se puede cambiar el tamaño de los píxeles con relativa facilidad. Al contrario que con otros software, no se remuetrea una capa raster, sino que se reproyecta todo un proyecto ('location') a la resolución deseada. Esto agiliza mucho el procesamiento de la información sin necesidad de generar infinidad de nuevos archivos con información parcialmente redundante. Se pulsa RETURN para acabar.
Finalmente se pide que se defina la resolución de los píxeles. Una cosa buena de GRASS es que se puede cambiar el tamaño de los píxeles con relativa facilidad. Al contrario que con otros software, no se remuetrea una capa raster, sino que se reproyecta todo un proyecto ('location') a la resolución deseada. Esto agiliza mucho el procesamiento de la información sin necesidad de generar infinidad de nuevos archivos con información parcialmente redundante. Se pulsa RETURN para acabar.
 Ya tenemos el proyecto creado. Ahora podemos comenzar GRASS desde la página de bienvenida (paso 1) seleccionando el 'database' donde has creado el nuevo 'location'. (/home/luis/grassdata), el 'location' que acabas de crear (Filabres) y el 'mapset' (PERMANENT). Pulsa 'Enter GRASS'.
Ya tenemos el proyecto creado. Ahora podemos comenzar GRASS desde la página de bienvenida (paso 1) seleccionando el 'database' donde has creado el nuevo 'location'. (/home/luis/grassdata), el 'location' que acabas de crear (Filabres) y el 'mapset' (PERMANENT). Pulsa 'Enter GRASS'.
 ¡Y ya podemos empezar a trabajar en GRASS!
¡Y ya podemos empezar a trabajar en GRASS!
Interfaz gráfica y consola de comandos ('shell')
En GRASS se puede trabajar de dos maneras (alternativa o simultáneamente): a través de la interfaz gráfica o a través de la consola de comandos ('shell'). Mientras que la primera es más intuitiva, la segunda da mayor versatilidad y potencia a la hora de programar funciones y procesos. Ambas opciones están disponibles todo el tiempo.
La interfaz gráfica consta de tres ventanas.
 La ventana principal es el administrador GIS (GIS Manager, izquierda). Desde esta ventana se pueden realizar la mayoría de las funciones relacionadas con el procesamiento de capas raster y vectorial, incluyendo la importación y exportación de capas en otros formatos. También se pueden modificar las características de configuración del 'location'.
La ventana principal es el administrador GIS (GIS Manager, izquierda). Desde esta ventana se pueden realizar la mayoría de las funciones relacionadas con el procesamiento de capas raster y vectorial, incluyendo la importación y exportación de capas en otros formatos. También se pueden modificar las características de configuración del 'location'.
Una segunda ventana (Output - GIS.m, arriba derecha) nos muestra los resultados de las operaciones realizadas a través del administrador GIS así como el código implementado (este código se puede usar directamente en la consola de comandos).
Finalmente hay una tercera ventana (Map Display X, abajo derecha) que muestra los mapas seleccionados en el administrador GIS. Otra de las ventajas de GRASS es que en vez de desplegar los mapas en la pantalla, envía el contenido del monitor a un archivo *.PNG por lo que ocupa menos memoria y es mucho más rápido a la hora de visualizar datos, especialmente cuando se trata de capas raster con una gran cantidad de información.
La consola de comandos ('shell') funciona de manera similar a como funcionan la mayoría de las aplicaciones en sistemas Unix. A través del 'shell' podemos obtener toda la funcionalidad que se obtiene a través de la interfaz gráfica de GRASS, pero además podemos programar scripts y conectar con otros software como R.
Importar datos a GRASS
Ya hemos creado nuestra 'location'. Ahora vamos a importar una capa raster a nuestro 'location' para trabajar con ella desde GRASS. GRASS es compatible con la mayoría de los formatos típicos utilizados en SIG y teledetección. En este caso importaremos una capa raster ascii de ArcGIS (GRID). Para ello vamos a:
File - Import raster map . ESRI grid
Alternativamente desde la consola de comandos podríamos haber escrito el comando:
r.in.arc
Cualquiera de las dos formas nos llevarían a la siguiente ventana, en dónde debemos de definir el archivo de entrada y el nombre del archivo de salida que se guardará directamente en nuestro 'location' y 'mapset' definidos. Una vez definidos los argumentos necesarios para ejecutar la función, pulsamos RUN.
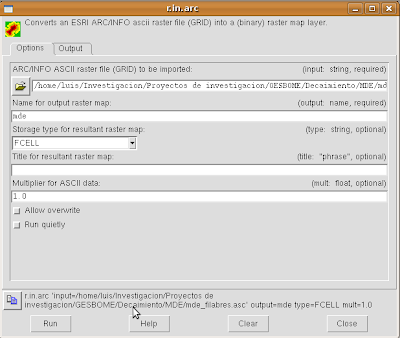
Como podemos observar en la parte inferior de la ventana aparece el código que ejecuta todo el proceso incluyendo la definición de los archivos de entrada y salida. Esta línea de texto puede ser ejecutada en la consola de comandos para realizar esta misma operación sin necesidad de tener que abrir un GUI (Graphic's User Interface).
Ahora ya podemos trabajar con esta capa raster en GRASS para realizar distintas operaciones. Para más información sobre cómo cargar capas en GRASS y visualizarlas se puede consultar el manual de Markus Neteler traducido por Guillermo Martínez.





























