La procesionaria del pino es una de las plagas más importantes de los pinares mediterráneos. Debe su nombre de "Procesionaria" a que se desplaza en grupo de forma alineada, a modo de procesión. En Andalucía, la procesionaria es un problema importante. Durante los últimos años, la Consejería de Medio Ambiente de la Junta de Andalucía ha estado recopilando información sobre el grado de afección de plantaciones y pinares autóctonos en toda Andalucía.
Aunque los datos que se muestran aquí son datos todavía incompletos, el objetivo es mostrar el potencial de R para el manejo, depuración y análisis de bases de datos extensas. R no sólo permite todo ésto, sino que además ofrece la posibilidad de trabajar con datos espaciales simulando las capacidades y potencialidades de SIG como ArcGIS o Idrisi.
Los datos están accesibles a través de un ftp, por lo que todo el código que se muestra a continuación se puede reproducir en R desde cualquier ordenador. El código se muestra en azul y letra Courier.
Empecemos cargando (e instalando si fuera esta la primera sesión) los paquetes con los que vamos a trabajar:
install.packages(c("sp", "maps", "maptools", "rgdal", "gpclib", "spatstat")) Aunque los datos que se muestran aquí son datos todavía incompletos, el objetivo es mostrar el potencial de R para el manejo, depuración y análisis de bases de datos extensas. R no sólo permite todo ésto, sino que además ofrece la posibilidad de trabajar con datos espaciales simulando las capacidades y potencialidades de SIG como ArcGIS o Idrisi.
Los datos están accesibles a través de un ftp, por lo que todo el código que se muestra a continuación se puede reproducir en R desde cualquier ordenador. El código se muestra en azul y letra Courier.
Empecemos cargando (e instalando si fuera esta la primera sesión) los paquetes con los que vamos a trabajar:
library(sp)
library(maps)
library(maptools)
library(rgdal)
library(gpclib)
library(spatstat)
Leemos ahora los datos de daños (en formato *.txt) por procesionaria en rodales forestales de Andalucía. Los datos, como ya dije, están en un ftp, por lo que podemos leerlos directamente desde R y salvarlos como un objeto de R al que llamaremos daño. Si los puertos de acceso no están habilitados puede ser que este código no funcione y no se pueda leer el archivo.
daño <- read.table(url( "ftp://fundacionfire.org:Fire1234@fundacionfire.org/data/Procesionaria/daños.txt"), header = T, sep = "\t")
str(daño)
'data.frame': 30550 obs. of 3 variables:
$ Rodal: Factor w/ 3382 levels "AL-000-001","AL-000-002",..
$ Fecha: Factor w/ 675 levels "10/20/2003","1/11/2003",..
$ Grado: int 0 0 0 0 0 0 0 0 0 0 ...
Es decir, tenemos un arreglo de datos (data.frame) con 30.550 observaciones que contienen información sobre: (1) el rodal (identificador del rodal en dónde se tomo el dato de campo), (2) la fecha (dd/mm/aa), y (3) el grado de afección (de 0 a 5, siendo 0 = ningún daño y 5 = daño severo). El primer paso que vamos a seguir es poner todas las fechas en año y eliminar toda la información referida al mes y al día en el que se hizo la observación. De paso, haremos algunas correcciones de años que están mal escritos (p.e. 0002 en vez de 2002).
daño$Fecha <- do.call("rbind", strsplit(as.character(daño$Fecha), split = "/"))[,3]daño$Fecha <- sub("0002", "2002", daño$Fecha)
daño$Fecha <- sub("0202", "2002", daño$Fecha)
daño$Fecha <- as.factor(daño$Fecha)
Ahora exploramos visualmente los datos.
plot(table(table(daño$Rodal)), xlab = "Número de datos por rodal", ylab = "Número de rodales", cex.lab = 1.3, cex.axis = 1.3)

Esta gráfica muestra el número de datos que tenemos por rodal. Como sólo hay una observación por año en cada rodal y tenemos información para el periodo 1992-2003 (ambos años inclusive), supuestamente deberíamos tener un máximo de 12 datos por rodal. Sin embargo vemos dos cosas en esta gráfica: (1) que muchos rodales no tienen información para el periodo completo, y (2) que para algunos rodales tenemos más datos de los esperados, llegando incluso a 81 datos por rodal. Esto indica que o bien los datos están repetidos o hay más información de la que cabría esperar. Vamos a ver si hay elementos duplicados y, en caso de que los haya, vamos a eliminarlos.
rodal.sel <- table(daño$Rodal)[table(daño$Rodal)>12]daño <- data.frame(daño, dup = paste(daño$Rodal, daño$Fecha, sep = ""))
daño <- daño[!duplicated(daño$dup), ]
daño <- daño[ , -4]
str(daño)
'data.frame': 30243 obs. of 3 variables:
$ Rodal: Factor w/ 3382 levels "AL-000-001","AL-000-002",..: 1 1 1 1 1 1 1 1 1 1 ...
$ Fecha: Factor w/ 12 levels "1992","1993",..: 12 11 10 9 8 7 6 5 4 3 ...
$ Grado: int 0 0 0 0 0 0 0 0 0 0 ...
Como se puede observar, ahora tenemos 30243 datos, frente a los 30550 que teníamos al principio. La diferencia entre estas dos cifras es la cantidad de elementos que estaban repetidos en la base de datos.
Lo que vamos a hacer ahora es separar los datos por años, de tal forma que tengamos una columna con el identificador del rodal y una columna por cada uno de los años para los cuales disponemos de datos. Esto podría ser tremendamente complicado con Excel ya que la información es altamente heterogénea y en cada año tenemos información sólo para unos rodales determinados. En R implica el uso de la función merge.
pt <- split(daño, daño$Fecha) a <- merge(pt[[1]], pt[[2]], by = "Rodal", all = T)
daño92_93 <- data.frame(Rodal = a$Rodal, Daño.1992 = a$Grado.x, Daño.1993 = a$Grado.y)
b <- merge(pt[[3]], pt[[4]], by = "Rodal", all = T)
daño94_95 <- data.frame(Rodal = b$Rodal, Daño.1994 = b$Grado.x, Daño.1995 = b$Grado.y)
c <- merge(pt[[5]], pt[[6]], by = "Rodal", all = T)
daño96_97 <- data.frame(Rodal = c$Rodal, Daño.1996 = c$Grado.x, Daño.1997 = c$Grado.y)
d <- merge(pt[[7]], pt[[8]], by = "Rodal", all = T)
daño98_99 <- data.frame(Rodal = d$Rodal, Daño.1998 = d$Grado.x, Daño.1999 = d$Grado.y)
e <- merge(pt[[9]], pt[[10]], by = "Rodal", all = T)
daño00_01 <- data.frame(Rodal = e$Rodal, Daño.2000 = e$Grado.x, Daño.2001 = e$Grado.y)
f <- merge(pt[[11]], pt[[12]], by = "Rodal", all = T)
daño02_03 <- data.frame(Rodal = f$Rodal, Daño.2002 = f$Grado.x, Daño.2003 = f$Grado.y)
daño <- merge(daño92_93, daño94_95, by = "Rodal", all = T) daño <- merge(daño, daño96_97, by = "Rodal", all = T)
daño <- merge(daño, daño98_99, by = "Rodal", all = T)
daño <- merge(daño, daño00_01, by = "Rodal", all = T)
daño <- merge(daño, daño02_03, by = "Rodal", all = T) str(daño)
'data.frame': 3382 obs. of 13 variables:
$ Rodal : Factor w/ 3382 levels "AL-000-001","AL-000-002"
$ Daño.1992: int 0 0 NA NA NA NA NA NA NA NA ...
$ Daño.1993: int 0 0 NA NA NA NA NA NA NA NA ...
$ Daño.1994: int 0 0 0 0 0 3 0 NA 2 0 ...
$ Daño.1995: int 0 0 0 0 0 1 0 NA 1 0 ...
$ Daño.1996: int 0 0 0 0 1 0 0 NA 0 0 ...
$ Daño.1997: int 0 0 0 0 2 1 0 NA 1 0 ...
$ Daño.1998: int 0 0 1 0 1 3 3 NA 2 1 ...
$ Daño.1999: int 0 0 0 0 0 1 0 NA 0 1 ...
$ Daño.2000: int 0 0 NA NA 0 1 0 NA 0 3 ...
$ Daño.2001: int 0 0 0 0 0 0 0 NA 1 0 ...
$ Daño.2002: int 0 0 0 0 0 0 0 0 0 0 ...
$ Daño.2003: int 0 0 0 0 0 0 0 0 0 0 ...
Lo que tenemos ahora es un arreglo de datos con 13 variables, la primera correspondiente al identificador y las doce siguientes haciendo referencia al año para el cual se tienen datos de daños. Hay un total de 3382 rodales para los cuales tenemos información. Sin embargo, como vimos antes, no para todos los rodales tenemos información de cada uno de los doce años del período 1992-2003. Vamos a ver cuántos rodales tienen valores faltantes (NA).
f1 <- function(x) sum(is.na(x))table(apply(daño, 1, f1))
0 1 2 3 4 5 6 7 8 9 10 11
313 216 1607 528 207 72 35 20 24 148 67 145
Es decir, que sólo tenemos información completa para todo el periodo para un total de 313 rodales. En 216 sólo falta un dato. En 1607 falta información de dos fechas. En 528 falta información de tres fechas, y así sucesivamente. Una vez más podemos observar que la información es bastante heterogénea.
El siguiente paso es asociar esta información a una capa vectorial de polígonos (formato shapefile *.shp) que representan los rodales de pinar en Andalucía. De esta manera podremos representar los datos en mapas. Para simplificar la lectura de datos, he incorporado esta capa y otra capa con las provincias de Andalucía en un objeto de R (*.Rob) que he subido al ftp.
load(url("ftp://fundacionfire.org:Fire1234@fundacionfire.org/data/Procesionaria/mapas.Rob"))
Esto importará a nuestra sesión de R dos objetos espaciales (SpatialPolygonsDataFrame) llamados mapa.rodales y andalucia. Lo primero que tenemos que ver es si los identificadores de los nombres de los rodales son iguales en la capa vectorial (mapa.rodales) y en nuestra base de datos (daño).
match(mapa.rodales@data$ID, daño$Rodal)Esto indica que no hay ninguna coincidencia. Si miramos más en detalle los nombres de los rodales en la base de datos, vemos que hay diferencias con respecto a la forma en la que se llaman en la capa vectorial. Así un rodal llamado AL-000-001 en la base de datos se denomina AL000001 en la capa vectorial. Vamos a solucionar ésto:
daño$Rodal <- gsub("-", "", daño$Rodal) Y ahora vamos a ver cuántos polígonos en la capa vectorial no tienen correspondencia con ninguno de los rodales en la base de datos y vice-versa.
sum(is.na(match(mapa.rodales@data$ID, daño$Rodal)))
[1] 625
sum(is.na(match(daño$Rodal, mapa.rodales@data$ID)))
[1] 59
Así que hay 625 polígonos para los cuáles no tenemos ni un sólo dato. ¿A qué es debido ésto? ¿Corresponden a otro tipo de vegetación? Igualmente hay 59 rodales en la base de datos que no tienen correspondencia con ninguno de los polígonos en la capa vectorial. Estos últimos datos no podremos usarlos porque no tienen representación espacial.
Lo siguiente es incorporar la información de la base de datos a la capa vectorial.
colnames(mapa.rodales@data)[1] <- "Rodal"
mapa.rodales@data <- merge(mapa.rodales@data, daño, by = "Rodal", all.x = T)
Y ahora ya sólo queda representar todos estos datos en mapas que generaremos para cada uno de los años del período de estudio. Es importante que antes de copiar este código establezcamos el directorio de trabajo. En Windows se puede hacer directamente desde Archivo (File). En Linux se puede hacer mediante el comando setwd().
mapa.rodales1 <- mapa.rodales
mapa.rodales1@data[, c(9:length(mapa.rodales1@data[1, ]))] <- mapa.rodales1@data[, c(9:length(mapa.rodales1@data[1, ]))] + 1
cols <- c("yellow", "orange1", "red1", "red2", "red3", "red4")
png(filename = "animation%02d.png", width = 800, height = 500)
for (i in 9:length(mapa.rodales@data[1, ])) {
plot(andalucia, axes = F, xlim = c(50000, 610000))
rrt <- mapa.rodales1@data[, i] plot(mapa.rodales, col = cols[rrt], border="transparent", las=1, add = T)
plot(andalucia, add = T)
axis(1, at = c(100000 + 0:4 * 125000), cex.axis = 1.2)
axis(2, at = c(3900000 + 0:4*100000), cex.axis = 1.2)
box()
legend("topleft", legend = c("Sin daño", "Daño bajo", "Daño medio bajo", "Daño medio alto", "Daño alto", "Daño severo"), fill = c("yellow", "orange1", "red1", "red2", "red3", "red4"), bty = "n", title = "Nivel de daño")
title(main = paste("Año", strsplit(colnames(mapa.rodales@data[i]), "\\.")[[1]][2]), cex.main = 2)
}
dev.off()
Esto va a crear doce mapas como el que se muestra a continuación.
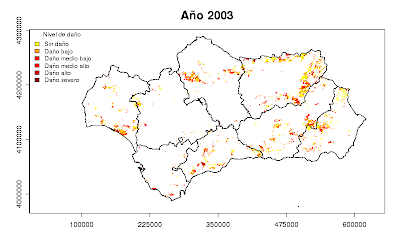
¡Y ahora la parte más interesante! Vamos a crear una animación con estas imágenes utilizando una aplicación de Java creada por Barry Rowlingson (http://www.maths.lancs.ac.uk/~rowlings/Chicas/DIY/), de la University of Lancaster.
El resultado final se puede observar en http://rsgis.ugr.es/sacacorchos/Animation/sample.html. Se puede regular la velocidad con la que se suceden las imágenes ... ¡Fantástico! ¿no?
El resultado final se puede observar en http://rsgis.ugr.es/sacacorchos/Animation/sample.html. Se puede regular la velocidad con la que se suceden las imágenes ... ¡Fantástico! ¿no?



No hay comentarios:
Publicar un comentario